鲫Kaiso基因cDNA的克隆和表达分析
鲫Kaiso基因cDNA的克隆和表达分析
陈海炎 黄万旭 罗 琛
(浙江大学生命科学学院, 杭州 310058)
在爪蟾和斑马鱼中, Kaiso是一种在整个基因组范围内与甲基化CpG序列特异性结合的转录抑制因子,在调控被甲基化基因表达的时间模式中起重要的作用。为深入研究DNA甲基化对我国重要养殖鱼类生殖和发育的影响, 我们克隆了鲫Kaiso基因的cDNA序列, 并对其时空表达模式进行了分析。该cDNA全长3145 bp, 5′-非翻译区132 bp, 3′-非翻译区1117 bp, 开放阅读框1896 bp, 编码631个氨基酸。鲫Kaiso蛋白与其他物种Kaiso蛋白的同源性分析表明, 与其他物种一样, 其 N端和C端分别有高保守性的BTB/POZ结构域和锌指结构域。整胚原位杂交结果显示, Kaiso mRNA在早期胚胎发育的各个时期均广泛表达, 信号均一, 但从尾芽期开始出现组织特异性表达差异。对不同发育阶段胚胎的实时定量PCR检测结果表明: 卵子中有高丰度的母源Kaiso mRNA存在; 在卵裂期至囊胚中期胚胎中Kaiso mRNA的丰度逐渐降低; 从囊胚中期至原肠早期都维持在最低水平状态; 原肠后期其表达水平又逐渐升高, 至尾芽期达到与未受精卵中相当的高水平后在器官发生期的整体水平又稍有下降。Kaiso mRNA丰度在胚胎发育早期的这种变化过程提示在卵裂期检测到的mRNA可能都是母源mRNA, 合子核Kaiso基因可能是在囊胚晚期后才开始转录。对成体不同组织的实时定量PCR检测结果表明Kaiso的表达存在明显的组织特异性差异, 在鲫肌肉、视网膜、心脏和脑中表达水平较高, 而在肾、胰、肝等器官中表达水平很低。Kaiso表达的时间和组织特异性提示其作为甲基化基因的转录抑制因子参与了胚胎和成体基因表达时空模式的调控。这些结果为进一步研究Kaiso和DNA甲基化修饰在鲫发育调控和遗传育种中的作用提供了基础资料。
鲫; Kaiso; 基因克隆; 表达时空模式
在脊椎动物中, DNA甲基化修饰与细胞的分化和发育[1,2], 基因的表达调节[3], 亲本印记[4], 染色质的稳定和X染色体失活[5]都有关, 并在沉默内源性反转录病毒, 免疫系统发育调节[6]和抑制同源重组[7]中发挥重要作用。通常认为, DNA甲基化是通过与甲基化的CpG二核苷酸特异结合蛋白的结合使特定位点染色质失活, 而抑制被甲基化基因的转录[8—10], 目前发现的这类蛋白主要有MeCP2、MBD1、MBD2、MBD4及Kaiso[11]。
Kaiso是BTB/POZ锌指结构转录因子家族的一员[12]。Kaiso最先是通过酵母双杂交实验, 用一种粘连蛋白p120-catenin作为诱饵将其筛选到的[13]。 Ruzov, et al.的研究证明了Kaiso是一种在整个基因组范围内与甲基化CpG序列特异性结合的转录抑制因子[14]。在爪蟾(Xenopus laevis)和斑马鱼(Danio rerio)中, Kaiso蛋白功能的缺失, 可以导致一些合子基因在囊胚中期转换之前提前表达, 出现部分细胞凋亡, 胚孔关闭延迟, 脑室和背轴结构发育严重畸形[14,15]。最近的研究证明鱼类Kaiso蛋白是抑制甲基化的母源no tail基因拷贝表达所必需的[16], 在维持双亲特异性甲基化基因的不对称表达中起重要作用。这些研究结果说明在鱼类和两栖类动物中, Kaiso在胚胎发育早期抑制被甲基化基因的表达和保障胚胎按正确的时空模式发育起重要的作用。
鲫(Carassius auratus)是我国广泛养殖的重要淡水经济鱼类和观赏鱼类, 并具有多种不同倍性和生殖方式的亚种, 是研究鱼类生殖控制和基因组进化的独特材料[17,18]。为开展DNA甲基化对重要养殖鱼类生殖、生长发育的调控作用及相关机制的研究,我们克隆了两性生殖鲫的Kaiso全长cDNA序列,并通过整胚原位杂交和实时定量PCR方法对其表达的时空模式进行了分析。
1 材料与方法
1.1 实验材料
本实验所用鲫为两性生殖的二倍体鲫 (Carassius auratus)。雌、雄鲫均于繁殖季节购自杭州市鱼类养殖场。雌、雄亲本在本实验室水族箱中分别养殖。交配前10h将雌、雄鲫鱼注射HCG(绒促性素, Human chorionic gonadotrophin, HCG; 宁波第二激素厂)进行人工催产后放入同一水族箱中。激素剂量为雌鱼每1 kg体重2000单位, 雄鱼剂量减半。发现产卵后挤取精子和卵子进行人工授精。胚胎置于(22±1)℃的环境发育。选取不同时期胚胎进行实验。肌肉、大脑、心脏、肾脏、胰脏、视网膜、肝脏等各成体组织均直接取自活体鲫鱼。
1.2 总RNA的抽提和cDNA第一链的获得
使用总RNA抽提试剂盒RNAgents Total RNA Isolation System (Promega)提取鲫胚胎不同发育时期及各成体组织的总RNA。抽提后的总RNA溶于无核酸酶水中置于−80℃冰箱中备用。以提取的总RNA为模板, 使用ClonTech公司的SMARTer™RACE cDNA Amplification的试剂盒分别合成5′RACE和3′RACE相应的cDNA文库。具体操作按试剂盒说明书进行。
1.3 鲫全长Kaiso mRNA的克隆
使用ClonTech公司的SMARTer™ RACE cDNA Amplification试剂盒。该试剂盒通过提供特定接头,无需进行传统的“去帽子”反应, 结合所设计的RACE特异性引物, 可以方便地对已知序列的5′端和3′端进行扩增。
通过比对已知的斑马鱼和爪蟾Kaiso基因序列,得到其相对保守的序列, 然后根据保守区域的序列设计并在上海生工合成了扩增鲫5′末端的一对引物: GK-5′-GSP: 5′CTTGTTGCAGTAATGGCACGGA3′(位置见图1)。依据试剂盒说明书进行5′RACE扩增, 扩增得到的序列送去上海生工测序, 测序后的结果经NCBI中的BLAST功能分析, 证明其为斑马鱼和爪蟾Kaiso的同源片段, 因此可确定此片段为鲫Kaiso mRNA 5′末端的序列。据5′RACE得到的序列, 设计并在上海生工合成了3′RACE特异性引物: GK-3′-GSP: 5′AGCAGTAACGGACCAACCAACG3′(位置见图1)。依据试剂盒说明书进行3′RACE扩增, 扩增得到的序列送去上海生工测序。 序列分析确定其为斑马鱼和爪蟾Kaiso3′端的同源片段后, 对所得的Kaiso mRNA的5′和3′片段进行拼接得到完整的金鱼Kaiso mRNA序列(图1)。
1.4 整胚原位杂交
根据1.3中所得到的鲫Kaiso mRNA序列, 设计并在上海生工合成了扩增鲫Kaiso 原位杂交探针的引物: Kaiso (+): 5′GCCATCGATATGTCGAAACTA AAGCTAATATCTGC3′; Kaiso (-): 5′ACCGAATTCT CAGTACGTCTCTGGTATAACAAAC3′。将扩出的序列克隆至pCS107 vector中, 用ClaI(Takara)酶切处理, 使重组质粒线性化, 以它为模板, 用T7 RNA聚合酶(Roche)和含有地高辛标记UTP的NTP(Promega)进行体外转录得到鲫Kaiso的反义RNA探针。整胚原位杂交的胚胎的固定和预处理、复水和探针的杂交、非特异性探针的洗脱、抗体的孵育以及显色等步骤和方法均按文献[19]所述进行。
1.5 荧光实时定量PCR
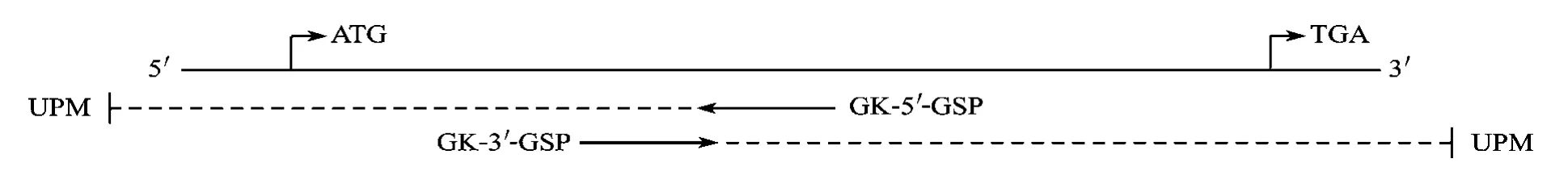
图1 克隆全长鲫Kaiso mRNA引物的位置示意图(GSP: 特异性引物; UPM: 通用引物)Fig. 1 Primer positions for cloning the full length of Kaiso mRNA (GSP: Gene-Specific Primer; UPM: Universal Primer A Mix)
运用荧光实时定量PCR的方法检测鲫胚胎发育不同时期及成体各组织中的转录水平。据1.3中克隆到的鲫Kaiso mRNA序列, 设计并在上海生工合成了实时定量PCR引物:GKaiso-RT(+): 5′TCAGGC TTTGGGAGTGACATT3′和GKaiso-RT(-): 5′CTTTG GAAAGGCCCGGTAAT3′。据鲫β-actin基因cDNA序列(GenBank 序列号: AB039726)设计并在上海生工合成了作为内参的实时定量引物: β-actin (+): 5′TCCCTTGCTCCTTCCACCA3′; β-actin (-): 5′GGAA GGGCCAGACTCATCGTA3′。用1.2中提取的总RNA为模板, 使用PrimeScript RT reagent Kit (Takara)合成mRNA第一条链, 具体操作按试剂盒说明书进行, 在此过程中使用无RNA酶的DNA酶消化可能存在于总RNA中的DNA干扰。实时定量PCR反应的体系为: 0.2 μL 2 mmol/μL的引物、1 μL逆转录产物、2.5 μL 2×SYBR Premix ExTaq(Takara)、1.1 μL灭菌水。反应条件为: 95℃, 10s; 95℃, 5s, 60℃, 20s, 40个循环。实时定量RT-PCR的反应及信息收集均在LightCycler 480 System(Roche)上进行。程序运行完成后进行溶解曲线分析以确定PCR产物是否专一,对每一样品分析重复三次以保证结果的可靠性。并使用β-actin作内参对照, 保证所有样品中RNA的量一致。目标基因的相对表达量按Livak和Schmittgen的方法计算[20]。据扩增曲线得到的Ct值(荧光信号达设定阈值时所经历的循环数)计算出目标基因Kaiso和对照基因β-actin Ct值的差异ΔCt; 然后以差异最大的样本作为参照样本, 计算出不同样品相对于参照样本基因的表达倍数2−ΔΔCt, 进而制作出相对定量图表。
2 结果
2.1 鲫Kaiso mRNA的克隆及序列分析
使用5′RACE和3′RACE技术, 得到鲫Kaiso全长mRNA的序列。鲫Kaiso mRNA全长序列的长度为3145 bp, 其中5′非编码区(5′-UTR)132 bp, 3′非编码区(3′-UTR)1117 bp, 开放阅读框(ORF)1896 bp, 编码631个氨基酸。序列已提交至GenBank (序列号: HM045515.1)。
2.2 鲫Kaiso蛋白结构的预测及进化关系分析
使用NCBI(http://www.ncbi.nlm.nih.gov/)中的Find Conserved Domains 功能, 并据推测的鲫Kaiso蛋白氨基酸序列(图2a), 将这一推测的氨基酸序列与斑马鱼、爪蟾、鸡(Gallus gallus)、小鼠(Mus musculus)、人(Homo sapiens)的Kaiso蛋白氨基酸序列进行相似性比较, 参照它们的蛋白结构, 预测得鲫Kaiso的蛋白结构(图2b)。鲫Kaiso蛋白也包括N端的BTB/POZ结构域和C端的三个锌指结构域(ZF1、ZF2、ZF3)(图2b)。

图2 推译的鲫Kaiso蛋白质氨基酸序列及结构域示意图Fig. 2 The putative amino acid sequence and the diagram of the putative conserved domains of Carassius auratus Kaiso protein
使用Jellyfish软件和NCBI (http: //www.ncbi.nlm. nih.gov/)中的BLAST, 将推译出的鲫Kaiso蛋白氨基酸序列与其他物种Kaiso蛋白氨基酸序列进行了同源性分析。结果表明鲫与斑马鱼、爪蟾、鸡、小鼠、人Kaiso蛋白氨基酸序列相似性分别为77%、41%、41%、42%、40.5% , 但它们N端BTB/POZ结构域和C端前两个锌指结构域(ZF1、ZF2)的相似性程度很高(表1)。据这几个物种的氨基酸序列, 用MEGA5.05软件Phylogeny程序中的Construct/Test Neighbor Joining Tree (s)方式构建出系统进化树(图3), 表明鲫首先与斑马鱼类聚, 再与爪蟾类聚, 再与鸡类聚, 最后与小鼠、人类聚。

表1 鲫与其他几种脊椎动物Kaiso蛋白质保守结构域(BTB/POZ、ZF1、ZF2、ZF3)氨基酸序列的同源性比较Tab. 1 Percent identity of amino acid sequence of Kaiso conserved domains (BTB/POZ, ZF1, ZF2 and ZF3) between goldfish and several other vertebrates
2.3 鲫Kaiso在胚胎发育过程中的时空表达模式和表达水平差异
为研究Kaiso在鲫早期胚胎发育过程中的时空表达模式, 以体外转录并用地高辛标记的Kaiso RNA 片段为探针, 对鲫不同发育时期的胚胎进行了整胚原位杂交实验。鲫不同时期胚胎原位杂交实验表明: Kaiso在早期胚胎发育阶段广泛表达, 信号均一, 但从尾芽期开始在胚胎前端的头部区域信号比胚胎后端区域显著增强(图4)。用实时定量PCR方法对未受精卵, 及不同发育时期胚胎进行Kaiso mRNA表达水平的相对定量分析结果显示, 卵子中存在高丰度的母源Kaiso mRNA, 在卵裂期及囊胚中期胚胎细胞中Kaiso mRNA的丰度逐渐降低, 从囊胚中期至原肠早期都维持在最低水平状态, 原肠后期其表达水平又逐渐升高, 至尾芽期达到与未受精卵中相当的高水平, 在器官发生期的整体水平又稍有下降但维持在较高水平(图5a)。

图3 根据鲫、斑马鱼、爪蟾、鸡、小鼠、人的Kaiso蛋白质氨基酸序列所构建的系统进化树Fig. 3 Phylogenetic tree of Kaiso amino acid sequences
2.4 鲫Kaiso在不同成体组织的表达分析

图4 鲫早期胚胎不同发育时期Kaiso的原位杂交图Fig. 4 In situ hybridization of Carassius auratus embryos
对成体不同组织的实时定量PCR检测结果表明Kaiso在鲫肌肉、脑、心脏、肾脏、胰脏、视网膜、肝脏等成体组织中均有表达, 但在鲫成体不同组织中的表达水平存在明显差异。在肌肉、视网膜、心脏和脑中表达水平较高, 而在肾、胰、肝等器官中表达水平较低(图5b)。
3 讨论
3.1 鲫Kaiso蛋白的结构
蛋白质氨基酸序列的保守性分析表明, 鲫Kaiso蛋白和其他已知的几种脊椎动物Kaiso蛋白一样, 在N端有保守的BTB/POZ结构域, C端有3个锌指结构域。对不同物种Kaiso蛋白的比较分析表明, 从哺乳类、鸟类、两栖类到最低等的脊椎动物鱼类, 其 N端的BTB/POZ结构域和C端的前两个锌指结构域的氨基酸序列是高度保守的, C端最后一个锌指结构域则变异比较大(表1)。功能分析表明, Kaiso蛋白是通过其N端的BTB/POZ结构域自二聚化[13], 或与其他蛋白如ZNF131发生异二聚化[21]而行使阻抑转录的作用。Kaiso与DNA的结合则主要通过其C端的锌指结构域, 但其第三个变异较大的锌指结构域并不是与甲基化的DNA结合所必需的, 其前两个锌指结构域就能充分介导与甲基化DNA的特异结合[15]。Kaiso N端的BTB/POZ结构域和C端前两个锌指结构域的高度保守性提示Kaiso蛋白与甲基化DNA特异结合后, 形成同二聚或者异二聚转录抑制复合体而抑制甲基化基因转录的特性对物种的生存至关重要。
3.2 鲫Kaiso表达的时空模式与发育调控的关系
在两栖类和鱼类中, Kaiso是一种与DNA上甲基化的CpG二核苷酸特异结合从而抑制被甲基化基因转录的转录抑制因子, 在抑制这些合子基因在胚胎囊胚中期转换之前表达中起关键作用, 而其与非甲基化DNA结合的功能不是鱼类和两栖类胚胎早期发育所需要的[14,15]。我们的实验结果表明鲫卵子中储存有大量母源Kaiso mRNA, 从卵裂期到囊胚中期其丰度逐渐下降, 从囊胚中期至原肠期维持在最低水平, 从原肠后期又开始升高, 至尾芽期达到最高水平。Kaiso mRNA丰度的在胚胎发育早期的这种变化过程提示在卵裂期检测到的mRNA可能都是母源mRNA, 合子核Kaiso基因可能是在囊胚晚期后才开始转录。
已有的研究证明在鱼类胚胎的早期发育过程中存在基因组DNA的去甲基化和再甲基化的重编程过程[22,33]。这一过程导致胚胎基因组DNA的甲基化水平在卵裂早期高, 去甲基化后水平降低, 再甲基化后水平又升高的变化过程。Kaiso mRNA表达水平变化也与斑马鱼中基因组DNA去甲基化和再甲基化的重编程所导致的基因组甲基化水平的变化一致, 但与参与甲基化重编程的鲫甲基化转移酶Dnmt1的情况正好相反[24]。这一结果提示Kaiso在鲫的整个胚胎发育过程中可能具有广泛的抑制甲基化基因表达的功能, 是调控这些基因按正确的时间模式表达的关键调控因子之一。
原位杂交结果显示, 在鲫胚胎发育的过程中, Kaiso在胚胎早期发育的各个时期均广泛表达, 且信号均一, 但从尾芽期开始在头部区域的表达显著强于在后端区域的表达。这些结果说明在器官发生阶段Kaiso的表达出现了组织特异性。成体不同组织中Kaiso的表达水平的实时定量PCR分析进一步证明了这种Kaiso表达的组织特异性: 在细胞分裂增殖活动较弱的肌肉、视网膜、心脏和脑等组织中的表达水平较高, 而在细胞分裂增殖旺盛的肾、胰、肝等器官中表达水平较低。鲫Kaiso基因的这种组织特异性表达式样提示其作为甲基化基因的转录抑制因子, 通过调控这些基因按正确的空间模式表达而参与了不同组织和器官功能的调控。

图5 鲫Kaiso基因的实时定量PCRFig. 5 Real-Time PCR detection of Carassius auratus Kaiso gene
[1] Ehrlich M. Expression of various genes is controlled by DNA methylation during mammalian development [J]. Journal of Cellular Biochemistry, 2003, 88(5): 899—910
[2] Li E. Chromatin modification and epigenetic reprogramming in mammalian development [J]. Nature Reviews Genetics, 2002, 3(9): 662—673
[3] Meehan R R, Stancheva I. DNA methylation and control of gene expression in vertebrate development [J]. Essays in Biochemistry, 2001, 37(5): 59—70
[4] Murphy S K, Jirtle R L. Imprinting evolution and the price of silence [J]. Bioessays, 2003, 25(6): 577—588
[5] Okamoto I, Otte A P, Allis C D, et al. Epigenetic dynamics of imprinted X inactivation during early mouse development [J]. Science, 2004, 303(5658): 644—649
[6] Fitzpatrick D R, Wilson C B. Methylation and demethylation in the regulation of genes, cells, and responses in the immune system [J]. Clinical Immunology, 2003, 109(1): 37—45
[7] Yoder J A, Soman N S, Verdine G L, et al. DNA (cytosine-5)-methyltransferase in mouse cells and tissues. Studies with a mechanism-based probe [J]. Journal of Molecular Biology, 1997, 270(3): 385—395
[8] Jones P L, Veenstra G J, Wade P A, et al. Methylated DNA and MeCP2 recruit histone deacetylase to repress transcription [J]. Nature Genetics, 1998, 19(2): 187—191
[9] Nan X, Ng H H, Johnson C A, et al. Transcriptional repression by the methyl-CpG-binding protein MeCP2 involves a histone deacetylase complex [J]. Nature, 1998, 393(6683): 386—389
[10] Wade P A, Gegonne A, Jones P L, et al. Mi-2 complex couples DNA methylation to chromatin remodelling and histone deacetylation [J]. Nature Genetics, 1999, 23(1): 62—66
[11] Hendrich B, Bird A. Identification and characterization of a family of mammalian methyl-CpG binding proteins [J]. Molecular and Cellular Biology, 1998, 18(11): 6538—6547
[12] Daniel J M., Spring C M, Crawford H C, et al. The p120 (ctn)-binding partner Kaiso is a bi-modal DNA-binding protein that recognizes both a sequence-specific consensus and methylated CpG dinucleotides [J]. Nucleic Acids Research, 2002, 30(13): 2911—2919
[13] Daniel J M, Reynolds A B. The catenin p120 (ctn) interacts with Kaiso, a novel BTB/POZ domain zinc finger transcription factor [J]. Molecular and Cellular Biology, 1999, 19(5): 3614—3623
[14] Ruzov A, Dunican D S, Prokhortchouk A, et al. Kaiso is a genome-wide repressor of transcription that is essential for amphibian development [J]. Development, 2004, 131(24): 6185—6194
[15] Ruzov A, Savitskaya E, Hackett J A, et al. The non-methylated DNA-binding function of Kaiso is not required in early Xenopus laevis development [J]. Development, 2009, 136(5): 729—738
[16] Ma S, Huang W, Zhang L, et al. Parent-specific methylation and genome evolution in primitive vertebrates [J]. Epigenetics, 2011, 6(12): 1471—1480
[17] Gui J F, Zhou L. Genetic basis and breeding application of clonal diversity and dual reproduction modes in polyploid Carassius auratus gibelio [J]. Science China Life Science, 2010, 53(4): 409—415
[18] Wang K Y, Chen X, Huang J L, et al. Effect of Ficus carica polysaccharides on non-specific immune response in crucain carp [J]. Acta Hydrobiologica Sinica, 2011, 35(4): 630—637 [汪开毓, 陈霞, 黄锦炉, 等. 无花果多糖对鲫鱼非特异性免疫功能的影响. 水生生物学报, 2011, 35(4): 630—637]
[19] Etsuor Y T M, Ken M, Yutaka H. Developmental staging in goldfish during the pre-gastrula stage [J]. Nippon Suisan Gakkaishi, 1999, 65(4): 709—717
[20] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C (T) Method [J]. Methods, 2001, 25(4): 402—408
[21] Donaldson N S, Nordgaard C L, Pierre C C, et al. Kaiso regulates Znf131-mediated transcriptional activation [J]. Experimental Cell Research, 2010, 316(10): 1692—1705
[22] Mhanni A A, McGowan R A. Global changes in genomic DNA methylation levels during early development of the zebrafish embryo [J]. Development Genes and Evolution, 2004, 214(8): 412—417
[23] MacKay A B, Mhanni A A, McGowan R A, et al. Immunological detection of changes in genomic DNA methylation during early zebrafish development [J]. Genome, 2007, 50(8): 778—785
[24] Zhang L, Xie B H, Zhang Q Y, et al. Molecular cloning and expression analysis of Dnmt1 in goldfish, Carassius auratus [J]. Acta Hydrobiologica Sinica, 2010, 34(2): 229—235 [张蕾, 谢冰花, 张琼宇, 等. 鲫鱼Dnmt1基因cDNA的克隆及表达分析. 水生生物学报, 2010, 34(2): 229—235]
声明:本站属公益性没有商业目的的网站,上列文章仅供个人学习参考。本站所发布文章为原创的均标注作者或来源,未经授权不得转载,许可转载的请注明出处。本站所载文章除原创外均来源于网络,如有未注明出处或标注错误或侵犯了您的合法权益,请及时联系我们!
欢迎关注本站(可搜索)"养鱼E线"微信公众帐号和微信视频号"养鱼一线"以及头条号"水花鱼@渔人刘文俊"!

